Read this article in Hindi to learn about:- 1. प्रोकेरियोट के प्री-प्राइमिंग व प्राइमिंग (Pre-Priming and Priming of Prokaryotes) 2. रेप्लिकेशन फोर्क की गति (Movement of Replication Fork) 3. लीडिंग स्ट्रैण्ड पर द्विगुणन (Replication on Leading Strand) 4. समापन (Termination) and Other Details.
द्विगुणक का प्रारम्भ तीन प्रक्रमों से होता है:
(i) Origin की पहचान ।
(ii) Duplex को खोलकर Single Strand का एक भाग तैयार करना तथा
ADVERTISEMENTS:
(iii) DNA B Protein (एक हैलीकेज) को पकड़ना ।
इन प्रक्रमों को कभी-कभी प्री-प्राइमिंग (Pre-Priming) (जो ऑरिजिन (Origin) पर होता है), प्राइमिंग (Priming) (जो प्रत्येक ओकाजाकी (Okazaki) टुकड़े की शुरूआत में होता है) तथा प्रीइनीशिएशन/इनीशिएशन कॉम्प्लैक्स (Pre-initiation/Initiation Complexes) का बनना भी कहते हैं । प्रारम्भन तरीके में विभिन्नताएं हो सकती हैं । यहाँ केवल E.Coli ori C का वर्णन किया जाएगा ।
प्रोकेरियोट के प्री-प्राइमिंग व प्राइमिंग (Pre-Priming and Priming of Prokaryotes):
Ori C से प्रारम्भन (Initation) में निम्नांकित चरण प्रयुक्त होते हैं:
(i) DNA A ATP कॉम्प्लैक्स 9bp उल्टे विलोम (Inverted Repeat) क्षेत्रों (R1, R2, R3, R4) को बाँधता है । यह DNA को एक 13bp क्रम (13-mers) के तीन सीधे Repeats क्षेत्र में खोलने में मदद करता है ।
ADVERTISEMENTS:
खोलने के लिए DNA-A-ATP कॉम्प्लैक्स के अलावा ऋणात्मक सुपरक्वॉइल्ड DNA (Negatively Supercoiled DNA) तथा Hu या IHE प्रोटीन की आवश्यकता होती है और 13-mer के दाहिने से शुरू होकर बायें की ओर खुलना प्रारम्भ होता है ।
(ii) ATP की उपस्थिति में Dna C की मदद से Dna B एकल स्ट्रैण्ड DNA (Single Stranded DNA) को स्थानान्तरित कर दिया जाता है ।
(iii) DNA B हेलिकेज (DNA B Helicase), ATP तथा SSB प्रोटीन (Single Strand Binding Protein) एवं DNA Gyrase के जुड़ने पर DNA को खोलता है । इससे प्री-प्राइमिंग कॉम्प्लैक्स (Pre-Priming Complex) का निर्माण होता है ।
Ori C से खुलता तथा द्विगुणन दोनों दिशाओं में होता है । SSB प्रोटीन का बँधना (Binding), एकल स्ट्रैण्ड (Single Stranded) क्षेत्रों में होता है । दो Dna B कॉम्फ्लैक्स, एक-एक स्ट्रैण्ड पर लोड (Load) हो जाते हैं ।
ADVERTISEMENTS:
(iv) खुलने के पश्चात् Dnag G प्राइमेज द्वारा RNA प्राइमर्स (Primers) का संश्लेषण होता है तथा DNA Pol III HE द्वारा (RNA) प्राइमर्स (Primers) का इलोंगेशन (Elongation) होता है ।
हेलिकेज (Helicase) तथा प्राइमेज (Primase) के गतिशील कॉम्प्लैक्स को प्राइमासोम (Primasome) कहते हैं । इसी तरह हेलिकेज, प्राइमेज व पॉलीमरेज (Helicase, Primase तथा polymerase) के चलित कॉम्प्लैक्स (Mobile Complex) को रेप्लिसोम (Replisome) कहते हैं । Ori C तथा Ori PI दोनों पर Dna A तथा प्रोटीन Dna B के स्थानान्तरण में मदद करती हैं ।
प्री-इनीशिएशन तथा इनीशिएशन कॉम्प्लैक्स का निर्माण (Formation of Pre-Initiation and Initiation Complex):
प्री-प्राइमिंग व प्राइमिंग (Pre-Priming and Priming) के बाद, DNA निर्माण के लिए, DNA Pol III का प्रयोग कर एक Initiation Complex बनता है ।
यह दो चरणों में बनता है:
(i) पहले चरण में स्लाइडिंग क्लैम्प (Sliding Clamp) (β2) DNA पर लोड (Load) हो जाता है । यह चरण ATP पर निर्भर करता है तथा मैचमेकर ϒ कॉम्प्लैक्स (Match Maker ϒ Complex ϒ2δ1δ’x1 Ψ1) द्वार उत्प्रेरित होता है । इस प्रकार प्री-इनीशिएशन कॉम्प्लैक्स (Pre-Initiation Complex) बनता है ।
(ii) दूसरे चरण में कोर (Core) एन्जाइम (αԑσ) β के साथ जुड़कर DNA से बँध जाता है और इस प्रकार इनीसिएशन कॉम्प्लैक्स (Initiation Complex) बनता है । ϒ कॉम्प्लैक्स (ϒ Complex) यद्यपि स्लाइडिंग क्लैम्प (Sliding Clamp) (ϒ) से अलग हो जाता है, फिर भी यह कोर तथा β2 से T2 की मदद से जुड़ा रहता है ।
T2 कोर, एन्जाइम तथा ϒ कॉम्प्लैक्स (ϒ Complex) के बीच पुल का कार्य करता है । अब स्लाइडिंग क्लैम्प (Sliding Clamp), डबल स्ट्रैण्ड DNA (Double Stranded DNA) के साथ कोर के सम्पर्क में रहती है, किन्तु ϒ Complex से दूर होती है ।
DNA Pol III HE लैंगिंग स्ट्रैण्ड (Lagging Strand) पर मल्टीपल इनीसिएशन (Multiple Initiation) में मदद करता है । द्विगुणन फोर्क (Replication Fork) पर बनने वाले प्रोटीन तथा एन्जाइम के कॉम्प्लैक्स (Complex) को भी रेप्लिसोम (Replisome) कहते हैं ।
DNA श्रृंखला का बढ़ना (Elongation of DNA Chain):
DNA द्विगुणन के आरम्भ के लिए Dna B Helicase की लोडिंग (Loading) तथा सक्रियता की आवश्यकता होती है । Dna B अन्य आवश्यक एन्जाइमों को निर्देशित (DNA G Primase तथा DNA Pol III HE) इलोंगेशन (Elongation) प्रक्रिया को आगे बढ़ती है । यहाँ संक्षेप में इलोंगेशन (Elongation) प्रक्रिया को समझाया गया है ।
DNA B प्रोटीन (DNA B Protein):
यह एक बड़ा हेलिकेज (Helicase) है । यह एक हेक्सामर (Hexamer) के रूप में होता है तथा DNA टेम्प्लेट (Template) का अनवाइन्ड (Unwind) (Ori C में 5’ → 3’ दिशा में) करता है । इसकी गति 500-1000 b/p/sec. होती है ।
अनवाइंडिंग (Unwinding) की दिशा के लिए DNAB को पॉलीमरेज (Polymerase) (Leading पर) से आगे लैनिंग स्ट्रैण्ड (Lagging Strand) पर उपस्थित होना पड़ता है । यह Dna G प्राइमेज (Primase) के द्वार प्राइमर (Primer) निर्माण को भी उत्तेजित करता है ।
चूंकि DNA हेलिकान (Helicane) केवल अनवाइण्डिंग (Unwinding) ही नहीं करता, बल्कि DNA के साथ ट्रान्सलोक्टे (Translocate) भी करता है । अत: इसे मोटर प्रोटीन (‘Motor Protein’) भी कहते हैं ।
DNAG प्राइमेज (DNAG Primase):
यह SSB लगे (Coated) एकल स्ट्रैण्ड (Single Stranded DNA) में एकल विशिष्ट ओलिगोन्यूक्लियोटाइड प्राइमर (Oligonucleotide Primer) का निर्माण करता है । प्राइमेज (Primase) के द्वारा निर्धारित क्रम 100 nt (nt = Nucleotide) लम्बा होता है ।
चूंकि प्राइमर (Primer) निर्माण के लिए Dna B का एक्टीवेशन (Activation) जरूरी है, अत: Dna B को मोबाइल प्रोमोटर (“Mobile Promoter”) भी कहते हैं ।
DNA पॉलीमरेज III होलोएन्जाइम (DNA Polymerase III Holoenzyme):
यह एक बड़ा मल्टी-प्रोटीन कॉम्प्लैक्स (Multi-Protein Complex) है, जो ई. कोली (E. Coli) में इलोंगेशन (Elongation) के लिए जिम्मेदार है । इसमें कम से कम 10 सबयूनिट (Sub-Units) होती हैं । प्रत्येक का विशिष्ट कार्य होता है ।
इस होलोएन्जाइम (Holoenzyme) में कोर-एन्जाइम (Core-Enzyme) तथा कुछ ”सहायक करक” होते है । यह T4 Phage तथा यूकैरियोट्स के एन्जाइम के समान कार्य करते हैं ।
इस होलोएन्जाइम (Holoenzyme) में β-सबयूनिट (B-Sub-Unit), अन्य यूनिट (Units) से दुगना होती है तथा कोशिका में यह पॉलीमरेज असेम्बली (Polymerase Assembly) से 300 गुना ज्यादा होती है । इसमें DNA को बाँधने के लिए एक स्लाइडिंग क्लैम्प (Sliding Clamp) होता है, जो T4 Gene 45 Protein तथा मानव PCNA के समान होता है ।
इनके अतिरिक्त DNA के Elongation में प्रयुक्त होने वाली अन्य प्रोटीन हैं:
(i) DNA गाइरेज (Gyrase)- यह एक टोपोआइसोमरेज (Topoisomerase) है । यह टोपोलॉजिक्ल (Topological) तनाव को दूर करता है तथा DNA की अनवाइंडिंग (Unwinding) से जुड़ा है ।
(ii) SSB Protein- यह एकल स्ट्रैण्ड बाइन्डिंग प्रोटीन (Single Strand Binding Proteins) हैं, जो कि अनवाउन्ड DNA (Unwound DNA) को स्थिर (Stabilize) करती हैं ।
(iii) RNase H- यह प्राइमर (Primer) को अलग करता है ।
(iv) DNA Pol I- यह RNA प्राइमर (Primer) के अलग होने से उत्पन्न रिक्त स्थानों से भरता है ।
(v) DNA लाइगेज (Ligase)- यह ओकाजाकी (Okazaki) टुकड़ों को जोड़कर लगातार स्ट्रैण्ड (Continuous Strand) बनाता है ।
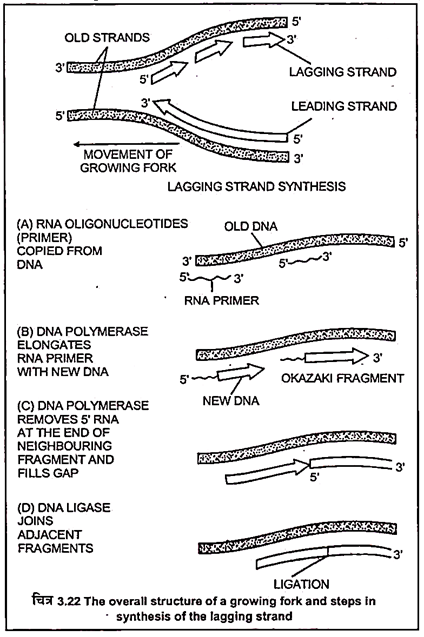
रेप्लिकेशन फोर्क की गति (Movement of Replication Fork):
आरम्भन के बाद इलोंगेशन (Elongation) के समय DNAB (5′ से 3′ दिशा में चलते वक्त), DNA डूप्लेक्स (Duplex) को खोलकर एक रेप्लिकेशन फोर्क (Replication Fork) बनाता है । DNA B युक्त स्ट्रैण्ड लैंगिग स्ट्रैण्ड (Lagging Strand) होता है ।
DNA प्राइमेज DNA B से जुड़ता है तथा मल्टीपल प्राइमर्स (Multiple Primers) (Lagging के लिए) तथा एकल प्राइमर (Single Primer) (Leading Strand के लिए) बनाता है । अब डाइमेरिक (Dimeric) DNA Pol III HE, दोनों स्ट्रैण्डों पर एक साथ जुड़ता है ।
लीडिंग स्टैण्ड (Leading Strand) से जुड़ा पॉलीमरेज (Polymerase),DNAB की दिशा में चलता है और उल्टी दिशा में नये स्ट्रैण्ड की नकल (Copy) करता जाता है । लैनिंग स्ट्रैण्ड (Lagging Strand) पर पॉलीमरेज (Polymerase) को DNAB की उल्टी दिशा में कार्य करना पड़ता है । Dna B, हेलिकेज (Helicase) तथा प्राइमेज (Primase) गतिविधियाँ प्राप्त करता है ।
इलोंगेशन (Elongation) में DNA Pol III HE तथा DNAB साथ-साथ कार्य करते हैं । यद्यपि दोनों ही फोर्क (Fork) से मजबूती से जुड़े रहते हैं । इसके विपरीत प्राइमेज (Primase), β2 तथा ϒ कॉम्प्लैक्स बिखरे रहते हैं तथा फोर्क (Fork) से पूरी तरह जुड़े नहीं रहते ।
लीडिंग स्ट्रैण्ड पर द्विगुणन (Replication on Leading Strand):
लीडिंग स्ट्रैण्ड (Leading Strand) पर केवल एक बार ही प्राइमर (Primer) लगता है । द्विदिशीय (Bidirectional) द्विगुणन में लीडिंग स्ट्रैण्ड (Leading Strands) की प्राइमिंग (Priming) के लिए किसी विशेष तकनीकी की आवश्यकता नहीं होती ।
इस स्ट्रैण्ड पर DNA पॉलीमरेज Polymerase भी अधिक सक्रिय रहता है । लीडिंग स्ट्रैण्ड (Leading Strand) पर निर्माण से एकल स्ट्रैण्ड DNA (Single Stranded DNA) का एक लूप (Loop) बनता है, जो लैगिंग (Lagging Strand) पर निर्माण (Synthesis) के लिए टेम्प्लेट (Template) का कार्य करता है ।
अनवाइण्डिंग (Unwinding) बढ़ने के साथ, यह लूप (Loop) भी बड़ा हो जाता है । इस लूप की लम्बाई कम से कम ओकाजा की टुकड़े (Okazaki Fragment) के जितना होती है ।
लैगिंग स्ट्रैण्ड पर टुकड़ों में द्विगुणन (Replication in Fragments on Lagging Strand):
Lagging Strand पर निर्माण कार्य अधिक जटिल होता है । साथ ही इस स्ट्रैण्ड (Strand) पर DNA पॉलीमरेज (Polymerase) भी कम सक्रिय होता है । प्राइमेज (Primase) की DNA B द्वारा सक्रिय बनाया जाता है, जिससे एक प्राइमर (Primer) (10-20 nt लम्बा) का निर्माण होता है ।
DNA Pol III HE, प्राइमेज से जुड़कर प्राइमेज (Primase) को फोर्क (Fork) के अलग करने का संकेत देता हैं । अब पॉलीमरेज (Polymerase), प्राइमर (Primers) की पहचान कर 2000-4000 तक ओकाजाकी (Okazaki) टुकड़ों को लैगिंग स्ट्रैण्ड (Lagging Strand) पर निर्माण करता है ।
प्रत्येक ओकाजाकी (Okazaki) टुकड़ा 1-2 kb लम्बा तथा निर्माण की गति 1 kb/sec. होती है । इसके लिए कोर पॉलीमरेज (Core Polymerase) की त्वरित पुनरावृत्ति (Recycling) जरूरी है ।
ओकाजाकी (Okazaki) टुकड़े के निर्माण के पश्चात् कोर तेजी से टूट जाती है । β2 छल्ला (Ring) पीछे छूट जाता है । कोर पुन: β2 क्लैम्प Clamp से जुड़कर आगे की नये प्राइमड की जगह (Primed Site) पर पहुँच जाता है और इस प्रकार नये ओकाजाकी (Okazaki) टुकड़े का निर्माण शुरू हो जाता है ।
DNA द्विगुणन का समापन (Termination of DNA Replication):
ई. कोली (E. Coli) में Ori C से दूर एक बड़ा समापन क्षेत्र (Termination Zone) होता है, जो कि रेप्लिकेशन फोर्क (Replication Fork) की गति को रोक देता है । E.Coli के क्रोमोसोम तथा कई प्लाज्मिड (Plasmids) में अनेक विशिष्ट क्रम होते हैं, जिन्हें ‘Ter Sites’ कहते हैं ।
इन्हें TBP (Ter Binding Proteins) तथा Tus Proteins बाँधती हैं । B.Subtilis में Tus के जैसी दूसरी प्रोटीन RTP- Replication Termination Protein होती है । E.Coli में समापन क्षेत्र (Termination Zone) में तीन Ter क्षेत्र (Sites) होती हैं- Ter A, Ter D तथा Ter E ।
ये तीनों काउन्टर क्लॉक के अनुसार (Counter-Clock Wise) फोर्क के लिए होती हैं, जबकि अन्य तीन (Ter B, टेर C तथा Ter F) क्लॉक के अनुसार (Clock Wise) Fork के लिए होती हैं । यह छ: Sites, Over Lapping तरीके से जुड़ी रहती है, जिससे कोई द्विगुणन मुक्त स्थान (Replication Free Gap) नहीं बचता ।
Tus-Ter Complex जो ‘Ter Site’ पर बनता है । द्विगुणन फोर्क (Replication Fork) को DNA हेलिकेज (DNA Helicase) तथा DNA B को रोक देता है । जब समापन क्षेत्र (Termination Zone) अलग हो जाता है, तो भी द्विगुणन (Replication) दोनों विपरीत फोर्क (Opposites Fork) के जुड़ने से रुक जाना यह बताता है कि समापन (Termination) के लिए समापन क्षेत्र (Termination Zone) अत्यावश्यक नहीं है ।
प्रोकेरियोट डी.एन.ए. पोलीमरेज (Prokaryotic DNA Polymerase):
DNA Polymerase-I:
यह Kornberg Polymerase कहलाता है । इसमें केवल दो Polypeptide Chain बना होता है, जिसमें Zn का परमाणु होता है । यह Damage DNA को Repair करने का कार्य करता । 5’→3′ की ओर यह 1000 Nucleotide प्रति मिनिट बना सकता है ।
इसके अलावा ये 3’→5′ Exonulease दर्शाता है तथा Mix Match Nucleotide को Remove करने का कार्य करता है, जो DNA की Strand में Polymerize के दौरान निर्मित हो गये थे । Mise. Match Nucleotide एक के बाद एक Remove कर दिये जाते हैं ।
Polymerase-I:
अत: Polymerase I, Prory Reading System का कार्य करता है । यह 5’→3’ Exonuclease Activity भी दर्शाता है । RNA Primere को Remove करने का कार्य इसी दिशा में होता है ।
Polymerase-II:
यह केवल एक Polypeptide Chain का बना होता है, जो 5’→3′ दिशा की और Polymerase दर्शाता है, जो E-Coli में 50, Nucleotide प्रति मिनिट बना सकता हैं । यह 5’→3’ दिशा में Exonuclear दर्शाता है ।
DNA Polymerase-III:
यह भी एक Polypeptide Chain का बना होता है । यह DNA को 5’→3’ Directional में बनाता हे । यह प्रमुख Polymerization Enzyme होता है, जो कि 15,000 Nucleotide प्रति मिनिट निर्मित कर सका है । यह कार्य यह 37°C पर E-Coli में करता है । अत: एक DNA की नई Strand पुरानी Strand पर निर्मित हो जाती है ।
प्रोकेरियोट में डी.एन.ए. द्विगुणन के चरण (Steps of DNA Replication in Prokaryotes):
Prokaryotes में Replication का प्रारंभ एक निश्चित Site से होता है, जो Bacteria के Chromosomes में Present होती है, जिसे Ori Site या Origin of Replication कहा जाता है, जो Bidirectional के लिए उत्तरदायी होती है ।
कोई भी DNA का अणु जो इस Sequence के अंतर्गत माना जाता है, Replication दर्शाता है तथा इस इकाई को Replication कहते हैं । Prokaryotic Cell के Genome में केवल एक Replicon पाया जाता है । इस प्रकार के Replication को Single Copy Replication कहते हैं ।
(1) ई. कोलाई में डी.एन.ए. द्विगुणन (DNA Replication in E. Coli):
Prokaryotic DNA गोलाकार, Double Stranded होता है । DNA Strand Antiparallel होती है । DNA की एक Strand जो 3’→5′ में चलती है, को DNA Polymerase III द्वारा Copy कर लिया जाता है 5’→3′ Directional में ।
इससे जो Complementary Strand प्राप्त होती है, उसे Leading Strand कहा जाता है तथा दूसरी Strand जो 5’→3′ में चलती है, Lagging Strand कहलाती है । यह Discontinuously निर्मित होती है, जिसकी वजह से DNA के Short Section का निर्माण होता है, जो 1,000 से 2,000 Nucleotides की होती है, उन्हें Okazaki Fragment कहते हैं ।
इन Fragment को DNA Ligase द्वारा बाद में जोड़ दिया जाता है, जिससे एक Continuous DNA Strand प्राप्त होती है । DNA Polymerase को अपने कार्य के लिए लगभग 10-Base’s लम्बी, RNA Primere को Sequence की आवश्यकता होती है, जो Chain के निर्माण के लिए उत्तरदायी होती है ।
(2) जीवाण्विक गुणसूत्र का पुनर्गुणन (Replication of Bacterial Chromosome):
अनुकूल स्थितियों में जीवाणु द्विभाजन द्वारा गुणन करते है । यह एक अति दुत विधि है । नवनिर्मित संतति जीवाणु से परिपक्व होने व दो में विभाजित होने में आवश्यक अवधि अथवा अनुक्रमिक पीढ़ियों के मध्य के समयान्तराल को पीढ़ी-काल कहा जाता है । यह 20 मिनट्स (जैसे- E-Coli से लेकर 15-20 घंटे) (जैसे- माएकोबैक्टीरियम ट्यूबरकूलोसिस) तक होता है ।
जीवाण्विक गुणसूत्र तो मीजोसोम में संलग्न रहते हुए पुनर्गुणन करते हैं । यहाँ पुनर्गुणन तो एकल उद्गम होता है, अर्थात् DNA के दो स्ट्रैण्ड्स एक बिन्दु पर पृथक होते है । पुनर्गुणन तो द्विदिशात्मकरूपेण अग्रसर होता है । DNA पुनर्गुणन के पूर्ण होने मर केन्द्राभ के दो संलग्नकारी बिन्दुओं के मध्य के क्षेत्र की वृद्धि द्वारा कोशिका की आकार वृद्धि आरम्भ हो जाती है ।
प्रक्रिया में मीजोसोम विभाजित होता है । संतति मीजोसोम्स की गति के साथ गुणसूत्र भी दूर गति करने लग जाते है । आरंभ में ये एकल Y-दुशाख के द्वार मीजोसोम्स से संलग्न होते है । नवीन संलग्न बिन्दु समीप ही उत्पन्न होता है ।